Die anhaltende Coronavirus-Pandemie stellt die Gesundheitssysteme weltweit vor große Herausforderungen. In verschiedenen Ländern wurden im Laufe der Pandemie neue SARS-CoV‑2 Varianten nachgewiesen, wobei besonders die „variants of concern (VOC)“ in den letzten Monaten an Bedeutung gewonnen haben. Zu diesen Varianten gehören besonders drei Virusvarianten die sich seit September 2020 im Südosten Großbritanniens (20I/501Y.V1, B.1.1.7), in Südafrika (20H/501Y.V2, B.1.351) und in Südamerika (20J/501Y.V3, P.1, B.1.1.28.1) ausbreiten. Diese VOC haben in bestimmten geographischen Regionen andere Virus-Varianten verdrängt und stehen in Verdacht, leichter übertragbar zu sein. Dafür sollen Mutationen im sogenannten Spike Protein (S‑Protein) verantwortlich sein.
Das Spike Protein ist für das Andocken der Viren an Atemwegszellen zuständig. Mutationen im S‑Protein können ebenfalls für die Unterscheidung der VOCs genutzt werden. Durch die erhöhte Übertragbarkeit könnten die VOC potenziell die Epidemie wieder verstärken und zu einer dritten Welle führen. Eine genaue Analyse der Ausbreitung der verschiedenen Virusvarianten ist daher dringend erforderlich, auch um weitere Fragestellungen wie z.B. die Wirksamkeit von Impfstoffen gegenüber verschiedenen Virusvarianten oder grundsätzliche Fragen zur Pathogenese der COVID-19 Erkrankung zu analysieren.
Aus diesem Grund wurden im Jahr 2020 mit Unterstützung des Bundesministeriums für Gesundheit in enger Zusammenarbeit von verschiedenen Forschungsgruppen am Forschungszentrum Borstel, Leibniz Lungenzentrum alle notwendigen Komponenten — von der „Drive thru“ Corona-Ambulanz zur Probengewinnung, über eine schnelle PCR-Diagnostik bis zur Genomsequenzierung von SARS-CoV‑2 mit verschiedenen „Next Generation Sequencing“ (NGS) Verfahren – aufgebaut und etabliert. Bei der NGS-Methode handelt es sich eine modernes Hochdurchsatzverfahren, das eine parallele und kostengünstige Sequenzierung des vollständigen Erbguts der Erreger ermöglicht.
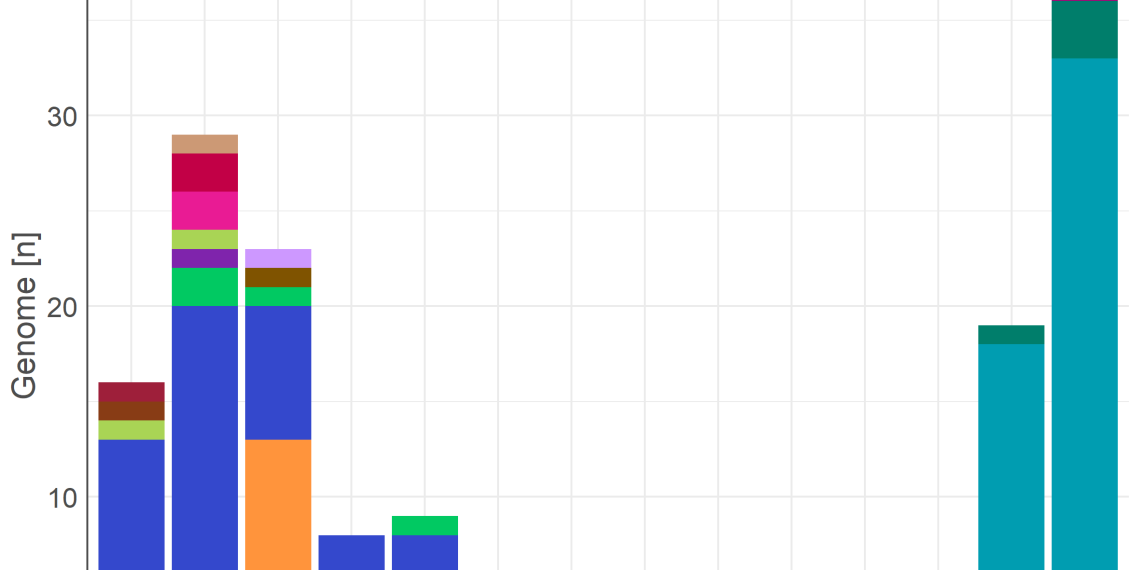
In den Jahren 2020 und 2021 wurden in der „Drive-Thru“ Corona-Ambulanz der Medizinischen Klinik Borstel Proben von mehr als 8000 Patienten auf COVID-19 getestet und in der Diagnostik mehr als 5000 SARS-CoV‑2 PCR Untersuchungen durchgeführt. Um eine prospektive, genombasierte Überwachung zu ermöglichen, wurde die Virus- Genomsequenzierung mit nachfolgender bioinformatischer Analyse, z.B. der Varianten-bestimmung, am FZB etabliert. Insgesamt wurden bisher 167 Genomsequenzierungen von Proben aus 2020 und 2021 durchgeführt. Die Viren wurden in die Haupttypen klassifiziert und Mutationen im S‑Protein bestimmt. Hierbei zeigte sich, dass die Viren aus dem Jahr 2020 hauptsächlich den Linien A und B1, die Viren aus dem Jahr 2021 hingegen hauptsächlich aus der Linie 20A.EU1 (B.1.177) (76%) stammten. Aus den 63 aktuellen Proben aus Schleswig-Holstein, gehörten 3 Proben zu der britischen VOC Linie 20I/501Y.V1 (B.1.1.7).
„Das Vorkommen verschiedener Virusvarianten aus den Jahren 2020 und 2021 zeigt sehr große Unterschiede und deutet eine hohe Variabilität in der Viruspopulation an, deren Bedeutung weiter untersucht werden muss. Die Übertragung von Viren der VOC Linie 20I/501Y.V1 (B.1.1.7) muss in Zukunft eng überwacht werden, um ihre weitere Ausbreitung besser kontrollieren zu können.“
— Prof. Stefan Niemann, Leiter der COVID-19-SH NET Studie am Forschungszentrum Borstel
Im Rahmen der durch das Ministerium für Bildung, Wissenschaft und Kultur des Landes Schleswig-Holstein geförderten COVID-19-SH NET Studie werden nun weitere Proben aus der „Drive Thru“ Ambulanz in Borstel und der Diagnostik am Universitätsklinikum Schleswig-Holstein sequenziert, um ein genaues Bild der Dynamik der Ausbreitung verschiedener Varianten in Zukunft zu ermöglichen. Übergeordnete Ziele von COVID-19-SH NET sind: 1) den Viren — Ausbruch auf genomischer Ebene weiter zu verfolgen und Mutationen im Virusgenom auf funktioneller Ebene zu untersuchen, 2) Risikofaktoren für Patient*innen mit schweren Verläufen besser zu definieren, 3) pathogenetische und immunologische Mechanismen, die die Schwere des Krankheitsverlaufs oder aber die Entwicklung von Schutzmechanismen beeinflussen, zu bestimmen.